A partir dessas informa��es j� conhecidas, uma pesquisa liderada por um grupo de professores dos departamentos de Bot�nica e de Morfologia do Instituto de Ci�ncias Biol�gicas (ICB) da Universidade Federal de Minas Gerais (UFMG) mostrou que as prote�nas ACE2 do indiv�duo, tamb�m variantes, podem alterar a intera��o do v�rus com a c�lula humana e impactar no potencial de transmiss�o do Sars-Cov-2, v�rus que transmite a COVID-19.
“Mostramos que algumas variantes do novo coronav�rus s�o mais sens�veis que outras varia��es gen�ticas humanas e ilustramos a evolu��o do v�rus em dire��o a uma maior afinidade com a ACE2 humana, e isso independentemente da varia��o na ACE2”, explica o biotecnologista e doutorando em Bioinform�tica na UFMG, Thiago Mendon�a dos Santos, orientado pelo professor Luiz-Eduardo Del-Bem, do departamento de Bot�nica do ICB.
Segundo o pesquisador, a afinidade tamb�m est� relacionada com a transmissibilidade. “A for�a da liga��o, ou seja, a afinidade da prote�na Spike com o ACE2 humano � um dos fatores que determina a facilidade com que o coronav�rus � transmitido de c�lula a c�lula”, ressalta.
O estudo demonstrou que as varia��es gen�ticas humanas na ACE2 influenciam na for�a da liga��o com as variantes do coronav�rus: P1 (Brasil), B.1.1.7 (Reino Unido) e B.1.351 (�frica do Sul).
O processo
O estudo foi feito a partir de an�lises in silico, ferramentas de bioinform�tica usadas para comparar um grande volume de dados. Em modelos tridimensionais, foram medidas a for�a de liga��o das duas prote�nas ligadas (ACE2-Spike) e a de suas variantes. “Tamb�m simulamos o complexo ACE2-Spike em condi��es fisiol�gicas, usando robustos processamentos computacionais”, afirma Thiago.
Tanto a prote�na Spike do v�rus quanto a ACE2 humana foram obtidas de bancos de dados p�blicos. As variantes ACE2 humanas foram selecionadas no banco de dados chamado de 1000 genomas, que tem mais de 2 mil genomas humanos sequenciados ao redor do mundo. Os processamentos de simula��o em condi��es fisiol�gicas foram feitos nos supercomputadores do Centro Nacional de Processamento de Alto Desempenho em S�o Paulo (Cenapad SP).
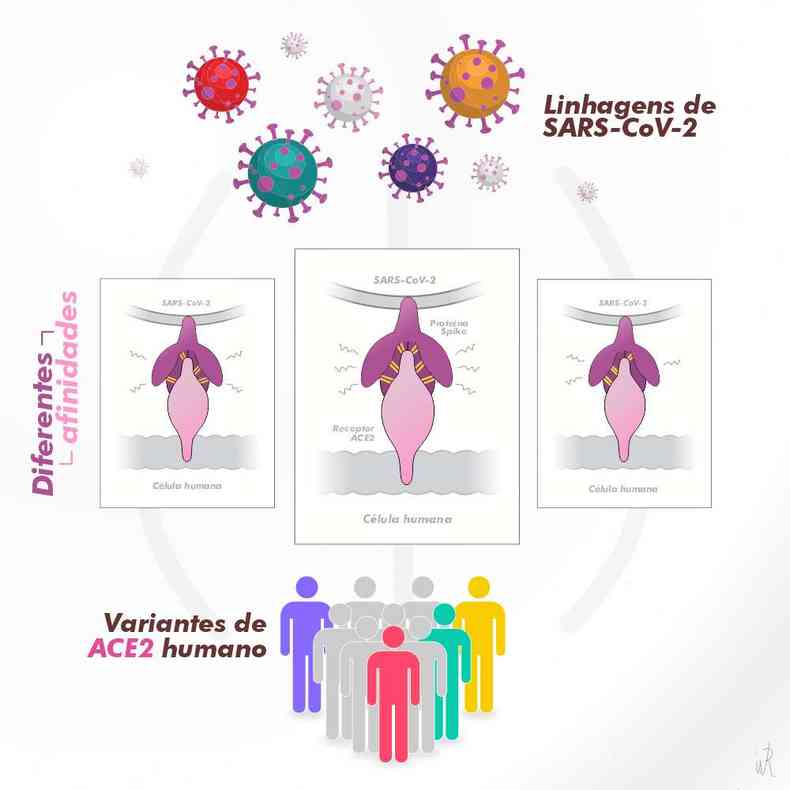
Dentre todos os dados analisados, os cientistas da UFMG optaram por selecionar 11 variantes de ACE2 que, somadas �s tr�s variantes de coronav�rus analisadas (alfa, gama e beta) e com a linhagem original de Wuhan, somam 48 complexos de prote�nas ligadas. A partir da� eles compararam os n�meros referentes a valores de afinidade encontrados em outros trabalhos publicados na �rea e identificaram os dados analisados.